4 ปัจจัย ทั่วโลกจับตา ‘โอไมครอน CH.1.1’ ที่จะมาแทนที่ BA.5 และ BA.2.75 พบในไทยแล้ว 9 ราย
ศูนย์จีโนมทางการแพทย์ รพ.รามาธิบดี โพสต์เพจเฟซบุ๊ก Center for Medical Genomics เรื่องการพบเจอ โอไมครอน ‘CH.1.1’ ที่จะมาแทนที่ BA.5 และ BA.2.75 พบในไทยแล้ว 9 ราย รวมถึง เรื่องการติดตามโอไมครอนที่อาจจะกลายพันธุ์ ก่อให้เกิดความรุนแรง ดีงนี้
“10 ประโยชน์”จากการศึกษาธรรมชาติการกลายพันธุ์ไวรัสโคโรนา 2019 ด้วยการถอดรหัสพันธุกรรมทั้งจีโนม
“4 ปัจจัย”ที่น่ากังวลต่อโอมิครอน “CH.1.1” หนึ่งในสมาชิก “ซุปโอไมครอน”, ลูกของ BA.2.75
“ปัญหา”เชื้อโควิดดื้อต่อยาต้านไวรัส (Anti-SARS-CoV-2 drugs) ที่ต้องเร่งแก้ไข
“องค์การอนามัยโลก” แถลงเตือนอาจเกิดสายพันธุ์ใหม่ที่รุนแรงหากการ์ดตก ถึงแม้ว่าประชากรโลกกว่า 90% จะมีภูมิคุ้มกันโควิด-19 แล้วก็ตาม

จับตา CH.1.1 ลูก BA.2.75ที่จะมาแทนที่ BA.5 พบในไทยแล้ว 9 ราย
การติดตามธรรมชาติการกลายพันธุ์ไวรัสโคโรนา 2019 ด้วยการถอดรหัสพันธุกรรมทั้งจีโนมจ ากตัวอย่างผู้ติดเชื้อทั้งในประเทศและระหว่างทวีป อัปโหลดไว้บนฐานข้อมูลโควิดโลก “กิสเสด(GISAID)” จำนวนกว่า “14 ล้านราย” ในขณะนี้ชี้ให้เห็นถึงการอ่อนกำลังลงของโอไมครอนรุ่นที่หนึ่ง “BA.4 และ BA.5” อย่างต่อเนื่อง
พร้อมเผยให้เห็นถึงการแข่งขัน ระหว่างสายพันธุ์โอไมครอนรุ่นที่สอง BA.2.75* (BN.1, CH.1.1), รุ่นที่สาม BQ.1*, และรุ่นลูกผสม XBB* และ XBF รวมถึงผลกระทบที่(อาจ)จะเกิดขึ้นในปีหน้า 2566 ตำแหน่งกลายพันธุ์บนโปรตีนหนามส่วนหนึ่ง ทำนายได้ว่าจะก่อความรุนแรงหรือไม่ ในขณะที่การกลายพันธุ์อีกหลายตำแหน่งไม่อาจคาดเดาว่ าจะลดหรือเพิ่มความรุนแรงในการติดเชื้อ
(ภาพ1)

สำหรับประเทศไทยหลายองค์กรทั้งภาครัฐ NGO และเอกชนได้ร่วมกันถอดรหัสพันธุกรรมไวรัสโคโรนา 2019 ทั้งจีโนมแล้วกว่า 32,938 ราย (3/12/2565) ได้ตรวจพบโอมิครอนสายพันธุ์ย่อยเกือบทุกสายพันธุ์ที่ทั่วโลกพบ กล่าวคือ BA.2.75* (BA.2.75, BA.2.75.2,BA.2.75.5), XBB* (XBB, XBB.1, XBB.1.1) BQ.1*(BQ.1, BQ.1.1), BN.1, BF.7, XBC, และ CH.1.1
(ภาพ2)
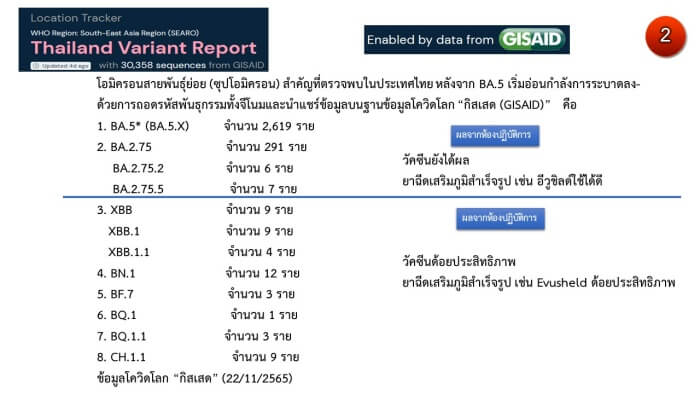
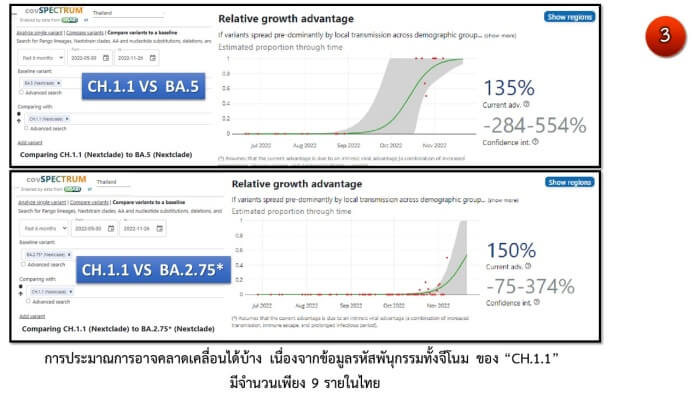
ที่ควรจับตาในไทยล่าสุดคือ “CH.1.1” ซึ่งเป็นรุ่นลูกของ BA.2.75 เนื่องจากในประเทศไทยโอไมครอน CH.1.1 มีความได้เปรียบในการเติบโต-แพร่ระบาด (relative growth advantage) เหนือกว่า BA.5 ประมาณ 135% และเหนือกว่า BA.2.75* ประมาณ 150% ทำให้มีแนวโน้มที่อาจระบาดมาแทนที่ BA.5 และ BA.2.75 ได้ ขณะนี้พบ“CH.1.1” ในไทยแล้ว 9 ราย
(ภาพ3)
4 ปัจจัยที่ผู้เชี่ยวชาญทั่วโลก เริ่มกังวล สายพันธุ์ย่อย โอไมครอน CH.1.1
มี 4 ปัจจัยที่ผู้เชี่ยวชาญทั่วโลกเริ่มกังวลกับโอไมครอนสายพันธุ์ย่อย CH.1.1 ซึ่งพบครั้งแรกในประเทศออสเตรียคือ
- พบตำแหน่งกลายพันธุ์บริเวณส่วนโปรตีนหนาม “P681R” ของโอไมครอน CH.1.1 เช่นเดียวกับที่พบในสายพันธุ์เดลตา ซึ่งอาจก่อให้เกิดการติดเชื้อมีอาการรุนแรงเช่นเดียวกับสายพันธุ์เดลตา เนื่องจากการทดสอบในห้องปฏิบัติการพบว่า ส่วนหนามที่กลายพันธุ์ไปสามารถจับกับผิวเซลล์จากอวัยวะอื่นได้หลากหลาย นอกเหนือไปจากเซลล์ปอด ทั้งยังก่อให้เกิดการหลอมรวมของเซลล์ที่อยู่ใกล้ชิดติดกันเป็นเซลล์ขนาดใหญ่ (fusogenicity) ดึงดูดให้ภูมิคุ้มกันของร่างกายเข้ามาทำลายเกิดอาการอักเสบที่รุนแรงขึ้น
- พบการกลายพันธุ์ในส่วนโปรตีนหนาม R346T, K444T, L452R, และ F486S ไปคล้ายกับโอไมครอนรุ่นที่ 3 “BQ.1.1”
- พบการระบาดของโอไมครอน CH.1.1 ได้อย่างรวดเร็ว โดยมีความได้เปรียบในการเติบโต-แพร่ระบาด (relative growth advantage) เหนือกว่า BQ.1.1 ที่ทั่วโลกถือว่าระบาดได้รวดเร็วที่สุดในปัจจุบันถึง 31% (ภาพ4)
- เกิดการกลายพันธุ์บนโปรตีนหนามในส่วนของ N-terminal domain (NTD), “NTD” บนส่วนหนามแหลมถือว่าเป็นบริเวณสำคัญ (hot spot) ที่แอนติบอดีที่ร่างกายสร้างขึ้นจากการกระตุ้นด้วยการฉีดวัคซีนหรือการติดเชื้อตามธรรมชาติ จะเข้าจับและทำลายไวรัส หากโปรตีนหนามบริเวณ “NTD” กลายพันธุ์ไปจะส่งเสริมให้ไวรัสหลบเลี่ยงภูมิคุ้มกันได้ดียิ่งขึ้น

โอไมครอน CH.1.1 พบแล้วหลายประเทศ รวมถึงประเทศไทย
ข้อมูลจากฐานข้อมูลรหัสพันธุกรรมโควิดโลก “กิสเสด” พบโอไมครอน CH.1.1 ในหลายประเทศโดยเริ่มต้นการระบาดมาจากประเทศออสเตรีย
- ออสเตรีย จำนวน 150 ราย ติดเป็น 0.209%
- ประเทศอังกฤษ จำนวน 137 ราย คิดเป็น 0.088%
- ออสเตรเลีย จำนวน 129 ราย คิดเป็น 0.246%
- สหรัฐ 74 ราย คิดเป็น 0.011%
- เดนมาร์ก จำนวน 54ราย คิดเป็น 0.060%
- ฮ่องกง จำนวน 27 ราย คิดเป็น 0.516%
- ญี่ปุ่น จำนวน 26 ราย คิดเป็น 0.017%
- เกาหลีใต้ จำนวน 20 ราย คิดเป็น 0.051%
- เนเธอร์แลนด์ จำนวน 17 ราย คิดเป็น 0.070%
- อินเดีย จำนวน 16 ราย คิดเป็น 0.047%
- ลิกเตนสไตน์ จำนวน 13 ราย คิดเป็น 3.171%
- อิสราเอล จำนวน 11 ราย คิดเป็น 0.020%
- สิงคโปร์ จำนวน 10 ราย คิดเป็น 0.110%
- ประเทศไทย จำนวน 9ราย คิดเป็น 0.188%
ฯลฯ

10 ประโยชน์ จากการถอดรหัสพันธุกรรมทั้งจีโนม
จากการติดตามธรรมชาติการกลายพันธุ์ไวรัสโคโรนา 2019 ด้วยการถอดรหัสพันธุกรรมทั้งจีโนมมาร่วม 3 ปี ทำให้ผู้เชี่ยวชาญทั่วโลกสรุปว่า
- จำเป็นต้องติดตามกลุ่มโอไมครอนสายพันธุ์ย่อย (omicron soup variants) ที่อุบัติขึ้นและระบาดพร้อมกันมาตั้งแต่ปลายเดือนตุลาคม 2565 เพราะหลายสายพันธุ์ย่อยมีการกลายพันธุ์โดยเฉพาะบริเวณโปรตีนหนามที่ “อาจ” ก่อให้เกิดการติดเชื้อรุนแรงต้องเข้ารักษาตัวในโรงพยาบาล และมีอัตราการเสียชีวิตเพิ่มขึ้น เช่น BA.2.75, BQ.1,BN.1, BF.7,CH.1.1 ฯลฯ (ภาพ1)
- ต้องกลับมาเฝ้าระวังไวรัสโคโรนา 2019 สายพันธุ์เก่าหลายสายพันธุ์ เช่น สายพันธุ์หวู่ฮั่นดั้งเดิม, อัลฟา, เบตา, เดลตา, แกมมา ฯลฯ ที่พบหมุนเวียนอยู่ ในตัวผู้ติดเชื้อบางกลุ่ม เสมือนแหล่งเก็บกัก (reservoir) เช่นผู้ที่มีภาวะภูมิคุ้มกันบกพร่อง ประกอบกับมีการผ่อนคลายมาตรการต่างๆ เช่น การ ลด ละ เลิกการสวมหน้ากากอนามัย การล้างมือ การเว้นระยะห่างทางกายภาพ และการฉีดวัคซีนเข็มกระตุ้น ทำให้สายพันธุ์ดั้งเดิมที่สูญหายไปแล้วอาจกลับมาปรากฏตัวได้อีกครั้ง
- ไม่ประมาทต่อการเกิดสายพันธุ์ลูกผสมระหว่างไวรัสตระกูลเก่า (หวู่ฮั่นดั้งเดิม, อัลฟา, เบตา, เดลตา) กับไวรัสตระกูลใหม่ (โอไมครอน) เช่น เดลตาครอน XBC (ภาพ5)
- ติดตามการเกิดสายพันธุ์ลูกผสมระหว่างสายพันธุ์ย่อยในตระกูลเดียวกัน (omicron soup variants) เช่น โอไมครอน XBB ซึ่งเป็นลูกผสมระหว่าง โอไมครอน BJ.1 x BA.2.75 (ภาพ6)

- พยายามควบคุมการแพร่ระบาดจากคนไปสู่สัตว์ อันจะก่อให้เกิดเป็นแหล่งเก็บกักโรค(reservoir) และเพาะบ่มให้เกิดไวรัสกลายพันธุ์ในสัตว์ที่ปราศจากแอนติบอดีมายับยั้ง และแพร่กลับมาติดต่อสู่คนอีกครั้ง ซึ่งเริ่มเห็นบ้างในกลุ่มกวางในแคนาดาและอเมริกาที่ติดเชื้อไวรัสโคโรนา 2019 จากคน จากนั้นพบมีการแพร่จากกวางกลับมาสู่คน (รายละเอียดเพิ่มเติม https://www.facebook.com/CMGrama/posts/pfbid0KdaqdbzhKHBqGV9t7Bx6pVTwjTz2NBMyuAWruEyfTp1zcHUcF3zc9tMARpYyPh1Zl)
- การตรวจวินิจฉัยด้วย ATK ยังใช้ได้ดีเนื่องจากเป็นการตรวจส่วนประกอบไวรัสหลายส่วนนอกเหนือไปจากโปรตีนหนาม
- ต้องหมั่นตรวจสอบประสิทธิภาพการตรวจจับไวรัสของชุดตรวจ PCR เนื่องจากไวรัสมีการกลายพันธุ์บนสายจีโนมอย่างต่อเนื่อง ทำให้ความไว (sensitivity) และความจำเพาะ (specificity) ในการตรวจจับไวรัสในสิ่งส่งตรวจของ PCR อาจลดลง
- ไม่ควรลดการสุ่มถอดรหัสพันธุกรรมไวรัสโคโรนา 2019 ทั้งจีโนม เพราะจะให้เราขาดข้อมูลของสายพันธุ์ที่กำลังระบาดในบ้านเราแบบเรียลไทม์ ซึ่งสำคัญอย่างมากต่อการควบคุม ป้องกัน และรักษาโรคโควิด
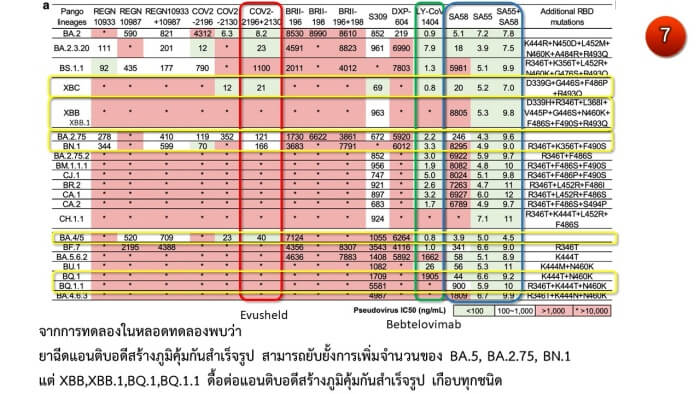
- เฝ้าระวังเชื้อดื้อยาทั้งจากยาเม็ดรับประทานและยาฉีด ด้วยการตรวจรหัสพันธุกรรม (genotyping & sequencing) เพื่อตรวจสอบสายพันธุ์และตำแหน่งกลายพันธุ์ที่เกี่ยวข้องกับการดื้อยา (anti-SARS-CoV-2 drugs) (ภาพ7)
- จัดลำดับโควิดสายพันธุ์ย่อยที่กำลังระบาด หรือมีแนวโน้มจะระบาดภายในประเทศ โดยอาศัยข้อมูลรหัสพันธุกรรม เพื่อเตรียมความพร้อมในการป้องกัน ด้านวัคซีนและยาต้านไวรัสให้สอดคล้องกับสายพันธุ์ที่ระบาด (ภาพ8)
WHO เตือน ประชากรโลกการ์ดตก อาจเกิดสายพันธุ์ใหม่ที่รุนแรง
เมื่อวันศุกร์ (2/12/2565) ที่ผ่านมา นพ. เทดรอส อัดฮานอม เกเบรเยซุส ผู้อำนวยการใหญ่องค์การอนามัยโลก ได้กล่าวเตือนว่า การผ่อนคลายมาตรการในการจัดการกับ COVID-19 ในปีนี้อาจสร้างเงื่อนไขให้เกิดไวรัสโคโรนา 2019 สายพันธุ์ใหม่ที่อันตรายร้ายแรงอุบัติขึ้นมาได้ ถึงแม้ว่าประชากรโลกกว่า 90% จะมีภูมิคุ้มกันโควิด-19 แล้วก็ตาม
https://www.youtube.com/watch?v=UW6W1UWqKoM
(ภาพ9)

ดังนั้นปลายปีนี้ต่อปีหน้า 2566 ยังคงต้องระมัดระวังสายพันธุ์ใหม่ที่ทำให้เกิดโรค และอาจเป็นอันตรายถึงชีวิต (COVID genomic surveillance) อย่างเข้มข้น พร้อมไปกับการเรียนรู้ผลกระทบระยะยาว ที่ประชาชนสัมผัสกับไวรัส SARS-CoV-2 อย่างต่อเนื่องมากว่า 3 ปีที่จะชัดเจนมากขึ้น รวมถึงผลกระทบของการติดเชื้อซ้ำ แม้จะฉีดวัคซีนป้องกัน (breakthrough infection) ที่เรียกว่า ” ไม่รุนแรง (mild)” รวมถึงประสิทธิภาพการฉีดวัคซีนเข็มกระตุ้น โดยเฉพาะวัคซีนสองสายพันธุ์ (bivalent vaccine)
อ่านข่าวเพิ่มเติม
- ‘หมอยง’ ไขข้อเท็จจริง คิดแบบง่ายๆ ‘เดลตาครอน’ จะมีโอกาส ระบาดในไทย หรือไม่?
- อย่าเพิ่งตกใจ! ศูนย์จีโนมฯ เผย ‘เดลตาคsอน’ จะมาแทนทื่ BA.5 และ BA.2.75 ไม่ง่าย เพราะเหตุนี้?
- สธ.ปรับแนวทางรักษา ‘โควิด’ ให้ ‘LAAB’ ในผู้ป่วยเบาหวาน หัวใจ หลอดเลือด และถุงลมโป่งพอง เริ่มวันนี้












